Miga M. & al. 2023
Deliry C. [2025] – Miga M. & al. 2023 - In : Odonates du Monde (Histoires Naturelles) (2004-[2025]) – Version 34365 du 01.08.2024. – odonates.net
|

Miga M. & al. 2023 - Characterization of the nearly complete mitochondrial genome of ochraceous darkies, Euphaea ochracea Selys, 1859 (Odonata: Zygoptera: Euphaeidae) and phylogenetic analysis. - Mitochondrial DNA Part B, Resources, 8 (2). - ONLINE (©© by)
Voir aussi Euphaea.
Résumé (traduction libre)
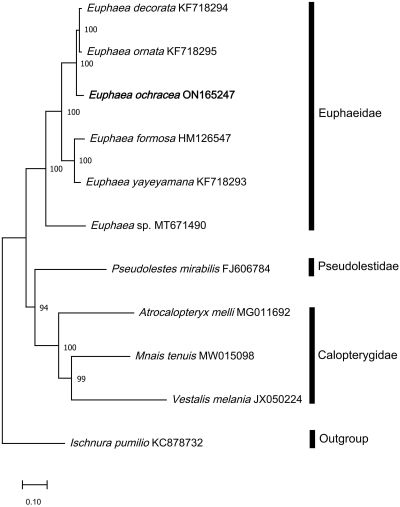
Dans la présente étude, le génome mitochondrial presque complet d'Euphaea ochracea a été décrit et sa position phylogénétique dans la famille des Euphaeidae a été analysée. Ici, nous avons récupéré 13 gènes codant pour des protéines, 22 ARN de transfert, 2 ARN ribosomiques et une région de contrôle partielle, ce qui donne une longueur de mitogénome de 15,545 pb. Tous les gènes codant pour des protéines ont été initiés par le codon typique ATN, sauf nad3 et nad1, qui utilise le codon TTG. Quatre gènes codant pour des protéines (cox1, cox2, cox3 et nad5) se terminent par un codon stop incomplet T, tandis que les autres se terminent par un codon TAA ou TAG. La région de l'espaceur intergénique, S5, est absente de ce mitogénome, soutenant l'absence de cette région comme un caractère spécifique chez les demoiselles. L'analyse phylogénétique a montré que le Euphaea ochracea nouvellement séquencé est phylogénétiquement plus proche de Euphaea ornata [et Euphaea decorata] avec une valeur de soutien élevée.
Abstract
In the present study, the nearly complete mitochondrial genome of Euphaea ochracea was described and its phylogenetic position in the family Euphaeidae was analyzed. Here, we recovered 13 protein-coding genes, 22 transfer RNAs, 2 ribosomal RNAs and a partial control region, resulting in a mitogenome length of 15,545bp. All protein-coding genes were initiated by the typical ATN codon except nad3 and nad1, which utilizes the TTG codon. Four protein-coding genes (cox1, cox2, cox3 and nad5) are terminated by an incomplete stop codon T, while others end with either a TAA or TAG codon. The intergenic spacer region, S5, is absent in this mitogenome, supporting the lack of this region as a specific character in damselflies. Phylogenetic analysis showed that the newly sequenced Euphaea ochracea is phylogenetically closer to Euphaea ornata [and Euphaea decorata] with a high support value.
|
| 🔍 - ©© by - Miga & al. (2023) : fig.3 - Analyse phylogénétique basée sur le maximum de vraisemblance de 11 séquences de mitogénome d'Odonates, y compris Euphaea ochracea nouvellement séquencé (Miga & al. 2023), en utilisant 13 gènes codant pour les protéines (PCG) concaténés. Toutes les séquences mitogéniques congénères des Euphaeidae ont été utilisées dans les analyses. Les valeurs de soutien nodal indiquent la valeur de soutien bootstrap du maximum de vraisemblance (BP). La nouvelle séquence d'Euphaea ochracea est mise en évidence en gras. Mnais tenuis (MW15098) (Wang et al. 2021 in Miga & al. 2023) ; Vestalis Melania (JX050224) (Chen et al. 2015 in Miga & al. 2023) ; Atrocalopteryx melli (MG011692) (Xu et al. 2018 in Miga & al. 2023) ; Euphaea decorata (KF718294), Euphaea ornata (KF718295), Euphaea yayeyamana (KF718293) (Cheng et al. 2018 in Miga & al. 2023) ; Euphaea sp. (MT671490) (Macher et al. 2020 in Miga & al. 2023) ; Euphaea ochracea (ON165247) (cette étude : Miga & al. 2023) ; Euphaea formosa (HM126547) (Lin et al. 2010 in Miga & al. 2023) ; Pseudolestes mirabilis (FJ606784) (non publié) ; Ischnura pumilio (KC878732) (Lorenzo-Carballa et al. 2014 in Miga & al. 2023). |

